Tuberculoseclusters in kaart gebracht door whole genome sequencing
Nederland heeft een belangrijke rol gespeeld in de ontwikkeling van DNA fingerprinting van Mycobacterium tuberculosis-isolaten om transmissie te volgen. Sinds 2016 wordt whole genome sequencing (WGS) toegepast in de tuberculosebestrijding, in eerste instantie in het kader van onderzoek, maar sinds 1 januari 2019 heeft WGS de vorige typeringsmethode (Variable Number of Tandem Repeats; VNTR) vervangen. WGS is een veel accuratere typeringsmethode, waardoor het aantal geclusterde patiënten afneemt (minder foutpositieve clustering) en kan nu veel efficiënter naar epidemiologische verbanden worden gezocht.In dit artikel beschrijven we de werking van WGS en de toepassing van WGS voor het monitoren van clustergroei en uitbraken. Daarnaast bespreken we de 4 grootste tuberculoseclusters in Nederland die gedurende de kwart eeuw van DNA fingerprinting zijn ontstaan en laten daarbij zien hoe grote tuberculose-uitbraken zeldzamer worden.Tot slot gaan we in op de afnemende expertise in het toepassen van WGS voor clusteronderzoek in de tuberculosebestrijding. Wij adviseren dan ook om de regionale aandachtfunctionarissen een sterkere rol te laten spelen in combinatie met een sterkere landelijke monitoring van transmissie en uitbraken in Nederland.
DNA fingerprinting
Al ruim 25 jaar wordt door het RIVM een DNA fingerprint gemaakt van elk M. tuberculosis-complex-isolaat in Nederland. Als deze fingerprints hetzelfde zijn (clusteren), bestaat er een kans dat patiënten elkaar geïnfecteerd hebben, of dat ze een gemeenschappelijke bron van infectie hebben. De DNA-typeringstechnieken zijn in 25 jaar veel veranderd en verbeterd. (1) De laatste state-of-the-art-techniek is WGS waarbij de gehele DNA-structuur van de tuberculosebacterie (4,4 miljoen basenparen) in kaart wordt gebracht.
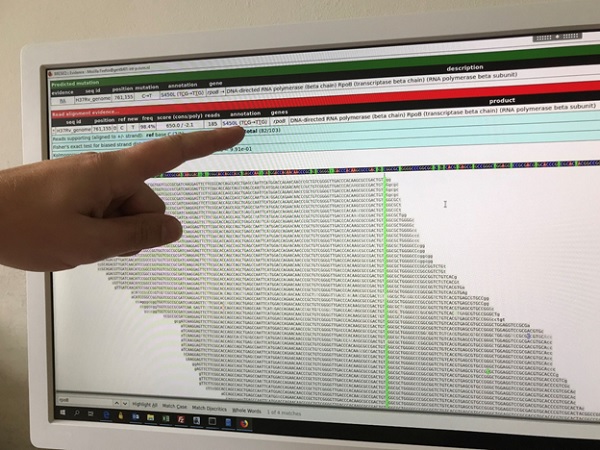
Foto. Schermbeeld met de volgorde (sequentie) van nucleotiden (A,T,C en G) in een klein stukje van het Mycobacterium-tuberculosisgenoom, in vergelijking met het referentiegenoom H37Rv. In het rpoB-gen is een C (cytosine) vervangen door T (thymine); de mutatie (single nucleotide polymorfisme) is groen gemarkeerd(Foto: Gerard de Vries)
In DNA komen de nucleotiden adenine (A), cytosine (C), guanine (G) en thymine (T) voor. In de WGS wordt de samenstelling van deze lange reeks (sequentie) van A’s, C’s, G’s en T’s in het genoom van de tuberculosebacterie vergeleken met een referentiegenoom (controlestam H37Rv) en met die van andere tuberculosebacteriën in het databestand (zie foto). In het kader van onderzoek is gedurende 2016-2018 van alle M. tuberculosis-complex-isolaten WGS uitgevoerd, waarover ook in het Infectieziekten Bulletin werd gerapporteerd. (2,3)
WGS-nummers en -clusters
Het RIVM kent na WGS-typering aan elk isolaat een nummer toe. Als er in het databestand al een (vrijwel) identiek WGS-patroon aanwezig is, krijgt het nieuwe isolaat hetzelfde nummer als deze bacterie. Dan ontstaat er een cluster of wordt een bestaand cluster met dit nieuwe isolaat/deze nieuwe patiënt uitgebreid. Voor clustering in de WGS wordt een definitie aangehouden van 12 of minder single nucleotide polymorfisme (SNP’s) verschil tussen 2 isolaten.De WGS-nummering is op 1 januari 2016 gestart met A001. Na de toekenning van nummer A999 is de nummering vervolgd met B001 totdat B999 wordt bereikt en daarna worden C-nummers ingevoerd, et cetera. De opeenvolgende WGS-aanduidingen zeggen dus niets over epidemiologische verbanden.
Patiënten 2016-2019
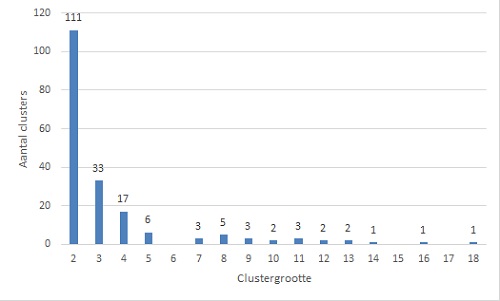
In de periode 2016-2019 zijn er 3.226 patiënten gemeld in het Nederlands Tuberculose Register (NTR): 887 in 2016, 783 in 2017, 797 in 2018 en 759 in 2019. (4,5) Totaal werd de diagnose bij 2.188 (68%) patiënten bevestigd met een kweek. Bij 1.850 (85%) van de meldingen was een ‘species identificatie’ (determinatie) van de bacterie in het NTR bekend. In 1.789 (97%) gevallen ging het om M. tuberculosis, 41x (2,2%) M. bovis, 15x (0,8%) M. africanum, 2x M. microti, 2x M. canettii en 1x M. bovis spp caprae. Bij 328 (15%) van de meldingen was M. tuberculosis complex ingevuld en 10 keer ‘onbekend’. Mogelijk was er geen speciesidentificatie uitgevoerd door het regionale of perifere microbiologische laboratorium, of was het resultaat niet bekend bij de GGD.Van 2.133 van de 2.188 (97%) van de patiënten met een met kweekonderzoek bevestigde diagnose was een WGS verricht van het M. tuberculosis-complex-isolaat. De WGS was bij 1.475 (69%) patiënten uniek en clusterde in 658 (31%) van de gevallen met die van 1 of meerdere andere patiënten. De patiënten met een clusterende bacterie vormden 190 clusters (figuur 1). De meeste clusters bestonden uit 2 of 3 patiënten (respectievelijk 111 en 33 clusters). In de 4 jaar dat WGS-typering werd uitgevoerd, waren er 10 clusters met meer dan 10 patiënten. Het grootste cluster (A016), dat al bekend was van de vroegere typeringen, bestond op grond van WGS uit 18 patiënten die voornamelijk uit Somalië en Eritrea kwamen (zie verder). Het één na grootste cluster in de WGS was een cluster van 16 patiënten in een uitbraak in 2016/2017 in een plaats in Gelderland. (6)
Figuur 1. Aantal en omvang van WGS-clusters, 2016-2019
‘n-1 methode’
In epidemiologische analyse van transmissie wordt de ‘n-1 methode’ gehanteerd. Hierbij wordt de eerste patiënt in het cluster als bron beschouwd (en niet meegeteld als geclusterd) en de rest in het cluster als vervolgpatiënten.(7) Volgens deze methode hadden 1.665 (1.475+190) (78%) patiënten een (initieel) uniek M. tuberculosis(-complex)-isolaat in WGS-typering en 465 (22%) een geclusterd isolaat.Er zijn een aantal voorwaarden bij het toepassen van deze methode; het sample moet voldoende representatief zijn (in onze studie werd 97% van de isolaten getypeerd) en de tijdsduur van typering moet voldoende lang zijn. (8) Bij de overgang naar een nieuwe typering wordt daarom in de tuberculosebestrijding eerst een databestand van 2 jaar aangemaakt om transmissie goed in kaart te brengen. Immers, mogelijke bronnen van verspreiding dienen in het bestand aanwezig te zijn en dat vergt bij tuberculose een aanzienlijke tijd. Er kan dus alleen een betrouwbare berekening gemaakt worden van het percentage vervolgpatiënten voor de jaren 2018 en 2019.Daarnaast is het belangrijk om een vaste vergelijkingsperiode te hanteren. In de tuberculosebestrijding wordt het begrip ‘recente clustering’ gehanteerd als de laatste patiënt in het cluster korter dan 24 maanden tevoren is gediagnosticeerd en ‘niet-recente clustering’ als het tijdsinterval met de laatste patiënt in het cluster meer is dan 2 jaar.In 2018 waren er 551 patiënten met een WGS-typering, waarvan er 147 (27%) een recent clusterende bacterie hadden; in 2019 was dat percentage 24% (118/491) (tabel 1).
Tabel 1. Classificatie van whole genome sequencing (WGS), 2018-2019
| WGS 2018 | Uniek | Recente clustering | Niet-recente clustering | Totaal |
| Uniek in 2018 | 382 | 382 | ||
| Nieuwe clusters | 20 | 26 | 46 | |
| Eerder bestaande clusters | 121 | 2 | 123 | |
| Totaal | 402 | 147 | 2 | 551 |
| WGS 2019 | Uniek | Recente clustering | Niet-recente clustering | Totaal |
| Uniek in 2019 | 338 | 338 | ||
| Nieuwe clusters | 13 | 15 | 28 | |
| Eerder bestaande clusters | 103 | 22 | 125 | |
| Totaal | 351 | 118 | 22 | 491 |
Clusteronderzoek
Als er recente WGS-clustering is van isolaten, doen sociaal verpleegkundigen tuberculosebestrijding van GGD’en onderzoek naar mogelijke epidemiologische links en transmissieroutes (clusteronderzoek). Deze resultaten zijn pas vele maanden na de diagnose bekend, waardoor alleen gegevens van patiënten die in 2018 zijn gemeld, bij het schrijven van dit artikel bekend zijn. Van de 147 recent geclusterde patiënten werd bij 121 (82%) een clusteronderzoek verricht en bij 26 (18%) niet. Redenen om geen clusteronderzoek te doen kunnen zijn: diagnosen die gesteld werden bij de binnenkomstscreening van immigranten of asielzoekers, diagnose bij een lopend contactonderzoek of dat de andere patiënt(en) in het cluster geen pulmonale vorm van tuberculose hadden. Bij clusteronderzoek werd bij 33 recent geclusterde patiënten (22%) een epidemiologisch verband aangetoond en bij 88 (60%) niet. De aangetoonde verbanden werden in 30 clusters gevonden (in 3 clusters waren 2 bevestigde verbanden).
Clustergroei in 2018
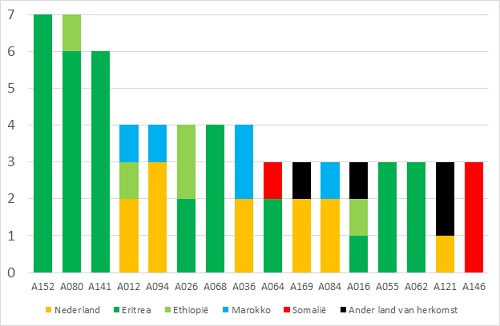
Figuur 2. Clusters met een toename van 3 of meer patiënten in 2018
Figuur 2 toont een overzicht van de 16 clusters die in 2018 met 3 of meer patiënten toenamen. Wat hierbij opvalt is dat de helft van de clusters (vrijwel) geheel bestaat uit patiënten met een Eritrese achtergrond. In 2018 was 1 van de 6 patiënten in Nederland (132 van 797) in Eritrea geboren. (4) Dit hangt samen met de grote instroom van Eritrese asielzoekers sinds 2014/2015, maar ook met het zeer hoge risico op tuberculose bij deze groep mensen. (9–11) Dat wordt nog eens verduidelijkt door WGS, want veel Eritreeërs hadden bij aankomst in Nederland tuberculose veroorzaakt door een clusterende bacterie in Nederland. Zo bestaat cluster A152 uit minderjarige, alleenstaande Eritrese vluchtelingen, waarvan 6 van de 7 korter dan 6 maanden in Nederland waren toen de diagnose werd gesteld. Hetzelfde is het geval bij cluster A080 (6 van de 7 patiënten korter dan 6 maanden in Nederland), cluster A141 (4 van de 6 korter dan 6 maanden in Nederland) en A064 (alle 3 korter dan 6 maanden in Nederland). WGS en epidemiologische informatie suggereren dat deze patiënten geïnfecteerd zijn door gemeenschappelijk bron(nen) tijdens de lange reis en het verblijf onder extreme omstandigheden in bijvoorbeeld Libische kampen. Dit is eerder beschreven in een groot Europees cluster van multiresistente tuberculose. (12) Daarentegen verbleven de Eritrese patiënten in clusters A026, A068, A016, A055 en A62 allemaal langer dan 6 maanden in Nederland, soms langer dan 2 jaar, toen de diagnose werd gesteld. Bij deze patiënten is het waarschijnlijk dat zij een reactivatie hadden van een latente infectie, opgelopen tijdens hun vlucht van een paar jaar eerder; het is ook mogelijk dat zij in Nederland zijn geïnfecteerd. (13)
Clustergroei in 2019
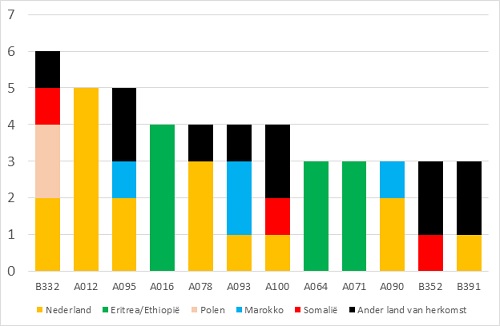
Figuur 3. Clusters met een toename van 3 of meer patiënten in 2019
Het aantal clusters dat in 2019 met 3 of meer patiënten groeide was kleiner dan in 2018; in totaal 12 clusters (figuur 3). Cluster B332 is een nieuw cluster met een aantal patiënten die verbleven in de daklozenopvang in Den Haag; dit cluster bestaat nu uit 7 patiënten. De eerste patiënt in dit cluster was in het najaar van 2016 gediagnosticeerd en van Poolse afkomst. In cluster A012 is, in de 4 jaar dat WGS-typering nu wordt toegepast, het aantal patiënten gestegen naar 14 (zie verder). Het is opmerkelijk dat de 3 clusters met een sterke toename in 2018 (A152, A080, A141), allemaal met Eritrese patiënten die recent in Nederland waren, maar gering groeiden in 2019, respectievelijk met 2, 2 en 1 patiënten.
Clustergroei sinds 1993
Om groeiende clusters en uitbraken in de tuberculosebestrijding goed te kunnen beoordelen, worden hieronder de DNA-fingerprintclusters besproken die in de jaren 1993-2019 met meer dan 100 patiënten groeiden. In deze periode zijn 3 DNA-typeringsmethodes toegepast: Restriction Fragment Length Polymorphism (RFLP) (1993-2008), VNTR (2009-2018) en WGS (2019). De M. tuberculosis-complex-isolaten 2004-2008 zijn behalve met RFLP later ook getypeerd met VNTR. Datzelfde is gebeurd met de isolaten 2016-2018 die naast een typering met VNTR ook met WGS zijn onderzocht. Omdat WGS een accuratere typeringsmethode is, zijn de WGS-resultaten voor de periode 2016-2018 in onderstaande clusterbeschrijvingen meegenomen en niet de gerapporteerde VNTR-resultaten. Totaal waren er 4 clusters met meer dan 100 patiënten, die we hieronder ieder apart zullen bespreken.
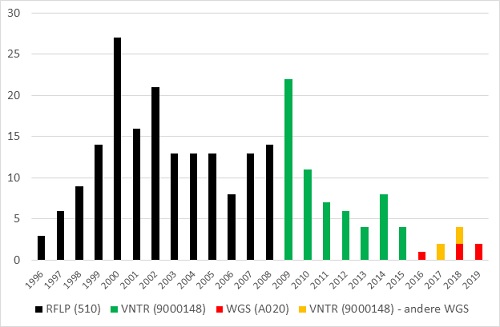
1. Cluster RFLP 510 – VNTR 9000148 – WGS A020
Figuur 4. Cluster RFLP 510 – VNTR 9000148 – WGS A020
Dit cluster met 256 patiënten is met afstand het grootste tuberculosecluster in Nederland (figuur 4). Tot dit cluster behoorden aanvankelijk alleen Kaapverdische patiënten. Een van de Kaapverdische patiënten in 1998 was drugsverslaafd. Door intensieve transmissie onder Rotterdamse daklozen en druggebruikers nam dit cluster eind jaren 90 van de vorige eeuw fors toe met mensen uit deze risicogroep en een aantal van hun familieleden en hulpverleners. Na 4 jaar gerichte radiologische screening van daklozen en druggebruikers werd het aandeel van deze groep sterk teruggebracht. (14) Het cluster groeide rond 2008-2009 opnieuw, met vooral Kaapverdische patiënten. (15)Tijdens de RFLP-typeringsperiode werd aan deze bacterie het nummer ‘510’ toegekend en groeide het cluster tot 171 patiënten. Bij de hertypering met VNTR (2004-2008) had 60/61 (98%) van deze RFLP 510-isolaten identieke VNTR-patronen (VNTR-nummer 9000148). Er waren echter ook 14 andere RFLP-isolaten die bij hertypering hetzelfde VNTR-patroon kregen (niet meegenomen in het clusteroverzicht). Bij de typering met zowel VNTR als WGS clusterden slechts 5 van 9 (56%) VNTR 9000148-isolaten in WGS (A020) en waren de overige 4 uniek. Mogelijk dat de VNTR-typering minder specifiek was voor deze specifieke bacterie, waardoor ook een aantal patiënten in de VNTR-typeringsperiode 2009-2015 (totaal 62) mogelijk ten onrechte aan dit cluster is toegewezen.
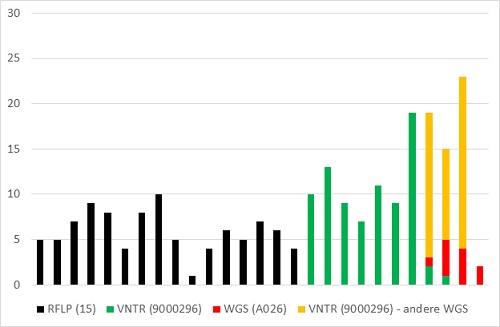
2. Cluster RFLP 15 – VNTR 9000296 – WGS A026
Figuur 5. Cluster RFLP 15 – VNTR 9000296 – WGS A026
Bij het op 1 na grootste cluster bestaan er al tientallen jaren twijfels of deze patiënten werkelijk een epidemiologische link hebben. Het RFLP-cluster 15 bestond voornamelijk uit Somalische patiënten, waarbij er zelden aanwijzingen waren voor transmissie tussen deze geclusterde patiënten. Tijdens de RFLP-typeringsperiode groeide het cluster gestaag tot 94 patiënten (figuur 5). Bij de hertypering met VNTR (2004-2008) bleek dat clustering vaak niet bleef bestaan in de VNTR-typering. Slechts 16/29 (55%) van deze RFLP 15-isolaten hadden identieke VNTR-patronen (cluster 9000296). De overige 13 isolaten hadden 10 verschillende VNTR-patronen (er was 1 cluster van 4 patiënten). Omgekeerd waren er 17 andere RFLP-isolaten die bij hertypering hetzelfde VNTR-patroon hadden (niet meegenomen in het clusteroverzicht). Er was dus in deze gevallen een lage concordantie (35%) tussen RFLP en VNTR. Bij de typering met zowel VNTR als WGS (50 isolaten) viel het VNTR-cluster volledig uiteen in 1 cluster van 9 patiënten (A026), 2 clusters van 4 patiënten (onder andere A064), 3 clusters van 3 patiënten, 3 clusters van 2 patiënten en 18 unieke WGS-isolaten. Het gebrekkig discriminerend vermogen van VNTR in clusters met veel Somalische patiënten werd ook in Denemarken waargenomen. (16)
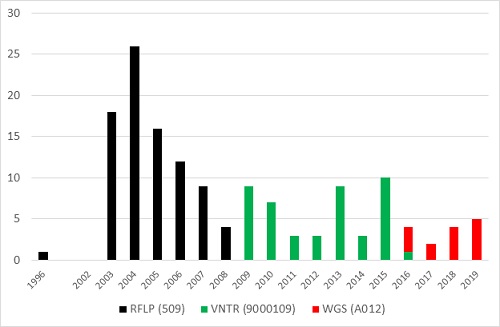
3. Cluster RFLP 509 – VNTR 9000109 – WGS A012
Figuur 6. Cluster RFLP 509 – VNTR 9000109 – WGS A012
Het derde grote cluster (n=145) ontstond door een uitbraak in 2013/2014 in de Rotterdamse cafés met vervolgens veel transmissie in de Eritrese gemeenschap. Tijdens de RFLP-typeringsmethode groeide het cluster tot 86 patiënten (figuur 6). Bij de hertypering had 61/65 (94%) van deze RFLP 509-isolaten identieke VNTR-patronen (cluster 9000109); er werden 4 andere RFLP-isolaten in dit cluster geplaatst. Opvallend is dat het cluster heel lang in Rotterdam geconcentreerd bleef: in de periode 2003-2014 woonde 85% (85/103) van de patiënten daar. Sinds 2015 groeide het cluster met patiënten die vooral in Noord-Holland en Flevoland woonden. Alle 14 VNTR-isolaten die ook met WGS werden getypeerd, clusterden in de WGS (cluster A012).
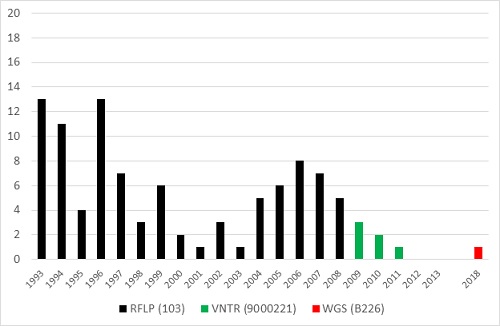
4. Cluster RFLP 103 – VNTR 9000221 – WGS B226
Figuur 7. Cluster RFLP 103 – VNTR 9000221 – WGS B226
Het vierde cluster met meer dan 100 patiënten (totaal 102) is bekend als het Harlingen-cluster (figuur 7). (17,18) Het cluster ontstond aan het begin van het DNA-fingerprinten in 1993, met ook enkele patiënten in 1992. De besmettingsbron was een Engelse lasser bij wie de ziekte een jaar lang onopgemerkt bleef. (17,19-22) Transmissie speelde zich vooral af in de cafés in Friesland. Er was een tweede verheffing in dit cluster in het eerste decennium van deze eeuw. Tijdens de RFLP-typeringsmethode groeide het cluster tot 95 patiënten. Bij de hertypering had 30/30 (100%) van deze RFLP 103-isolaten identieke VNTR-patronen (cluster 9000221). De laatste 5 jaar is nog slechts 1 patiënt aan dit cluster toegevoegd en het cluster lijkt nu definitief uit te doven.
Sterkere regionale ondersteuning en landelijke rol in monitoring clustergroei en uitbraken
DNA fingerprinting van M.-tuberculosis-complex-isolaten ondersteunt al tientallen jaren de tuberculosebestrijding in Nederland. De typeringstechnieken zijn steeds verder verfijnd, tot de huidige WGS-typering waarbij het gehele genoom in kaart wordt gebracht. De 3 verschillende typeringen hebben in die jaren een aantal grote uitbraken aangetoond, waarvan de staart soms nog aanwezig is of in andere gevallen is uitgedoofd. In 3 van de 4 clusters met meer dan 100 patiënten waren de verschillende DNA-fingerprintgegevens consistent en was er werkelijk sprake van grote uitbraken, hetgeen bevestigd werd door epidemiologisch onderzoek. In het andere grote cluster waren de 2 voorgaande typeringen inferieur en zijn deze patiënten ten onrechte in 1 cluster geplaatst. Met de nieuwe typeringsmethode wordt dit laatste cluster opgesplitst in verschillende andere clusters, hetgeen overeenkomt met de epidemiologische kenmerken van deze patiënten.
WGS en aanvullend epidemiologisch onderzoek laten zien dat recente transmissie steeds minder voorkomt en tuberculose in Nederland goed onder controle is. Bij de huidige patiënten is de infectie vaak opgelopen in het buitenland - bij immigranten vaak voor de komst naar Nederland in hun land van herkomst en bij asielzoekers waarschijnlijk ook tijdens de lange vluchtroute - of is de ziekte het gevolg van re-activatie van oude infecties die jaren eerder in Nederland zijn opgelopen. Met WGS is nu een kwaliteitsinstrument voorhanden dat zeer nauwkeurig de aan- of afwezigheid van transmissie en uitbraken kan vaststellen en monitoren. De (actieve) ziektegevallen zijn echter de ene kant van de transmissiemedaille. De andere kant is de ‘voorwaartse’ transmissie: GGD’en voeren contactonderzoek uit naar aanleiding van de melding van een patiënt met besmettelijke tuberculose, waarbij contacten opgespoord, onderzocht en, indien van toepassing, preventief worden behandeld. Het NTR verzamelt ook deze informatie, zodat we nu 2 meetinstrumenten in handen hebben die uitvoering geven aan de aanbeveling van het Nationaal plan tuberculosebestrijding 2016-2020 om transmissie in Nederland te monitoren en terug te dringen. (23,24)
De afname van het aantal patiënten en het afnemende aantal WGS-geclusterde patiënten en uitbraken, heeft ook een keerzijde, namelijk dat sociaal verpleegkundigen tuberculosebestrijding in de loop der tijd over veel minder expertise zullen beschikken om op de juiste manier clusteronderzoek te kunnen uitvoeren. Concentratie van expertise op landelijk en regionaal niveau lijkt daarom noodzakelijk, alsmede continue educatie over en ondersteuning bij clusteronderzoek. Recent hebben daarom de 4 regionale expertisecentra (REC’s) waarin GGD’en ten aanzien van tuberculosebestrijding samenwerken, een sociaal verpleegkundige tuberculosebestrijding aangewezen als aandachtsfunctionaris voor DNA fingerprinting. Het RIVM zal de clustergroei nauwkeurig blijven monitoren en GGD’en actief benaderen als clusters met 4 of meer patiënten toenemen in een periode van 12 maanden.
Auteurs
G. de Vries (1), K. van Beurden (1), M. Kamst-van Agterveld (1), K. Rebel (2), Y. Veulings (3), D. van Soolingen (1)
- Centrum Infectieziektebestrijding, RIVM, Bilthoven
- Afdeling tuberculosebestrijding, GGD Amsterdam, Amsterdam
- Afdeling tuberculosebestrijding, GGD Gelderland-Zuid, Nijmegen
Correspondentie
Literatuur
- Van Soolingen D, de Vries G, van Hunen R, Kamst M, de Beer J. Epidemiologische typering van Mycobacterium tuberculosis - van faagtypering tot Whole Genome Sequencing in twee decennia. Nederlands Tijdschrift voor Medische Microbiologie. 2014;22:55–60.
- Jajou R, Neeling A de, Hunen R van, Vries G de, Schimmel H, Mulder A, et al. Epidemiological links between tuberculosis cases identified twice as efficiently by whole genome sequencing than conventional molecular typing: A population-based study. PLoS ONE. 2018;13(4):e0195413.
- Jajou R, van Soolingen, D. Whole genome sequencing van Mycobacterium tuberculosis. Infectieziekten Bulletin [Internet]. 2019;30(6). Available from: https://magazines.rivm.nl/2019/11/infectieziekten-bulletin-0/whole-genom...
- Tuberculose in Nederland 2018 - Surveillancerapport inclusief rapportage monitoring van interventies. Bilthoven: RIVM; 2019.
- Tuberculose Kerncijfers Nederland 2019 [Internet]. 2020. Available from: https://www.rivm.nl/tuberculose/surveillance/stand-van-zaken
- Binnenlandse signalen. Contactonderzoek tuberculose door de GGD Noord- en Oost Gelderland. Infectieziekten Bulletin. 2017;28:368.
- Small PM, Hopewell PC, Singh SP, Paz A, Parsonnet J, Ruston DC, et al. The epidemiology of tuberculosis in San Francisco. A population-based study using conventional and molecular methods. N Engl J Med. 1994;330:1703–9.
- Glynn JR, Vynnycky E, Fine PE. Influence of sampling on estimates of clustering and recent transmission of Mycobacterium tuberculosis derived from DNA fingerprinting techniques. Am J Epidemiol. 1999;149:366–71.
- De Vries G, Gerritsen RF, van Burg JL, Erkens CGM, van Hest NAH, Schimmel HJ, et al. Tuberculose bij de twee meest voorkomende groepen asielzoekers in Nederland. [Tuberculosis among asylum seekers in the Netherlands]. Ned Tijdschr Geneeskd. 2016;160:D51.
- Wolters B, Aartsma Y, van Hest R, Schimmel H, van Hunen R, de Vries G. Toename tuberculose bij alleenstaande minderjarige asielzoekers uit Eritrea. Infectieziekten Bulletin. 2019;30(1).
- Van den Boogaard J, Slump E, Schimmel HJ, van der Hoek W, van den Hof S, de Vries G. High Incidence of Active Tuberculosis in Asylum Seekers from Eritrea and Somalia in the First 5 Years after Arrival in the Netherlands. Emerging Infect Dis. 2020 Apr;26(4):675–81.
- Walker TM, Merker M, Knoblauch AM, Helbling P, Schoch OD, Werf MJ van der, et al. A cluster of multidrug-resistant Mycobacterium tuberculosis among patients arriving in Europe from the Horn of Africa: a molecular epidemiological study. Lancet Infect Dis. 2018;18(4):431–40.
- Gerritsen RF, van Hunen R, van Soolingen D, de Vries G. “Whole genome sequencing” bevestigt twee tbc-uitbraken in Limburg. Tegen de Tuberculose. 2018;114(1):6–8.
- De Vries G, van Hest RA, Richardus JH. Impact of Mobile Radiographic Screening on Tuberculosis among Drug Users and Homeless Persons. Am J Respir Crit Care Med. 2007;176:201–7.
- De Vries G. Wat is de toegevoegde waarde van DNA fingerprinting voor de grootstedelijke tuberculosebestrijding? Infectieziekten Bulletin. 2008;19:207–10.
- Jajou R, de Neeling A, Rasmussen EM, Norman A, Mulder A, van Hunen R, et al. A Predominant Variable-Number Tandem-Repeat Cluster of Mycobacterium tuberculosis Isolates among Asylum Seekers in the Netherlands and Denmark, Deciphered by Whole-Genome Sequencing. J Clin Microbiol. 2018;56(2).
- Kiers A, Drost AP, van Soolingen D, Veen J. Use of DNA fingerprinting in international source case finding during a large outbreak of tuberculosis in The Netherlands. The International Journal of Tuberculosis and Lung Disease. 1997 Jun 1;1(3):239–45.
- Schürch AC, Kremer K, Daviena O, Kiers A, Boeree MJ, Siezen RJ, et al. High-Resolution Typing by Integration of Genome Sequencing Data in a Large Tuberculosis Cluster. J Clin Microbiol. 2010 Sep 1;48(9):3403–6.
- Kiers A. Tuberculose-explosie in Harlingen. Tegen de Tuberculose. 1993;89(3):62.
- Drost A. Opsporing verzocht. Bronopsporing in Harlingen. Tegen de Tuberculose. 1994;90(1):8–11.
- Kiers A, Drost AP, van Soolingen D, Veen J. Grensoverschrijdende bronopsporing bij tuberculose door DNA ’fingerprint’-techniek. Ned Tijdschr Geneeskd. 1996;140(46):2290–3.
- Kiers A. De verspreiding van cluster 103 vanuit Harlingen. Tegen de Tuberculose. 2007;103(3):8–11.
- De Vries G, van Dissel J, van Soolingen D. Measuring tuberculosis transmission in low-incidence countries. Lancet Respir Med. 2018;6(4):e13.
- Nationaal plan tuberculosebestrijding 2016-2020. Op weg naar eliminatie. Bilthoven: RIVM; 2016.
Infectieziekten Bulletin, jaargang 31, nummer 2, november 2020