Surveillance van Shigatoxineproducerende Escherichia coli in Nederland, 2019

Sinds 1999 wordt Shigatoxineproducerende Escherichia coli (STEC) in Nederland gevolgd met surveillance, bestaande uit een meldingsplicht en laboratoriumsurveillance. In 2019 werden 459 patiënten met een STEC-infectie gemeld, waarvan 35 patiënten met een STEC O157-infectie, 59 met een STEC non-O157-infectie, waaronder 17 STEC O26, en 365 patiënten met STEC zonder verdere typering. Het hemolytisch-uremisch syndroom (HUS) werd gemeld voor 22 patiënten (2x O157, 4x O26, 1x O113 en 15 zonder informatie over O-typering). STEC O157 en STEC O26 blijven de belangrijkste serotypes in Nederland. Voor het vaststellen van mogelijke bronnen van infectie onderzoekt Wageningen Food Safety Research (WFSR) in opdracht van de Nederlandse Voedsel- en Warenautoriteit (NVWA) monsters van voedsel en landbouwhuisdieren op STEC. In de niet-humane isolaten werden voornamelijk STEC O146, O113, O100 en O8 aangetroffen. De overstap naar WGS voor alle STEC-isolaten heeft de clusterdetectie versneld en verbeterd. Er werden in 2019 meerdere WGS-clusters van patiëntisolaten gezien, maar geen met zowel humane als voedselisolaten. Het steeds lagere aantal meldingen van STEC-patiënten waarbij ook een patiëntisolaat werd ingestuurd door het betreffende (regionale) laboratorium (26%), vormt een grote bedreiging voor de surveillance omdat er dan geen goed landelijk beeld is van circulerende typen en er geen adequate uitbraakdetectie/-onderzoek kan worden uitgevoerd.
STEC is een bacterie die maag-darmklachten kan veroorzaken met symptomen variërend van diarree tot hemorragische colitis en het hemolytisch-uremisch syndroom (HUS). (1) STEC is een zoönose met herkauwers, voornamelijk runderen, als belangrijkste reservoir. (2) STEC-infecties zijn vaak het gevolg van de consumptie van besmet voedsel, maar transmissie via het milieu lijkt ook belangrijk. (2, 3) Acute STEC-infecties zijn meldingsplichtig onder de Wet publieke gezondheid (Wpg), vanwege de ernst van de ziekte, vooral bij kleine kinderen en ouderen, en het risico op grootschalige uitbraken. In dit artikel presenteren we de resultaten van de surveillance voor het jaar 2019.
Methoden
Sinds juli 2016 worden patiënten alleen gemeld als met PCR (polymerase chain reaction) 1) het shigatoxinegen1-(stx1) en het escV-gen of E. coli attaching-and-effacing gen (eae-gen) is aangetoond, 2) het stx2-gen is aangetoond, 3) STEC met kweek is aangetoond. Verder worden alleen de acute infecties gemeld: maximaal 21 dagen tussen eerste ziektedag en afnamedatum, en tenminste diarree, braken en/of bloed in de ontlasting. De meldingen worden gedaan in Osiris. In Osiris is een extra vragenlijst opgenomen voor informatie over risicofactoren. Deze vragenlijst wordt alleen ingevuld als de infectie in Nederland is opgelopen of als dit onbekend is.
De laboratoria kunnen STEC-kweken opsturen naar het RIVM. Daar worden alle STEC-verdachte isolaten met PCR getest op de aanwezigheid van de belangrijkste virulentiegenen (stx-genen, eae-gen en het EHEC-hemolysinegen (EHEC-hly-gen)). Als er virulentiegenen aangetoond kunnen worden, wordt het isolaat geanalyseerd met whole-genome sequencing (WGS). De WGS-data worden gebruikt voor in silico serotypering en clusterdetectie met core-genome multi-locus sequence typing (cgMLST).
De resultaten van de surveillance over 2019 worden gerapporteerd met een onderverdeling naar STEC O157, STEC non-O157 en niet-getypeerde infecties. De meldingen van niet-getypeerde infecties hebben betrekking op patiënten die na RIVM-onderzoek niet positief bleken voor STEC of op patiënten van wie geen isolaat was ingestuurd.
De Nederlandse Voedsel- en Warenautoriteit (NVWA) wordt ingeschakeld als het vermoeden bestaat dat de bron van besmetting levensmiddelen zijn of contact met landbouwhuisdieren. Zij neemt monsters van de besmettingsbron of uit de omgeving voor onderzoek naar STEC en eventueel het bepalen van serotypes. Daarnaast onderzoekt de NVWA in het kader van diverse monitoringsprojecten jaarlijks relevante producten op de aanwezigheid van STEC. Het laboratoriumonderzoek wordt uitgevoerd door Wageningen Food Safety Research (WFSR).
STEC wordt voornamelijk gevonden in rundvlees. De focus van het STEC-onderzoek door WFSR/NVWA ligt daarom op vlees: vers vlees, gehakt vlees en vleesbereidingen (vers vlees waarin levensmiddelen, kruiden of additieven zijn toegevoegd). De hoeveelheid die onderzocht wordt is 25 gram product van de genomen monsters uit de detailhandel of 5 monsters van 1 partij uit de groothandel. De gevonden stammen worden gecontroleerd op de aanwezigheid van stx-genen, getest op aanhechtingsgenen (eae, aggR+aaiC) en het serotype wordt bepaald. De methoden bij het onderzoek van de NVWA zijn gebaseerd op de ISO-methode 13136 en de diverse typerings PCRs van het EURL-VTEC. De methode voor het aantonen van stx2f is gebaseerd op Derzelle et al. (4) Van de voedselisolaten zijn ook WGS-data (cgMLST) beschikbaar, zodat een vergelijking met de patiëntisolaten gemaakt kan worden.
Resultaten
Patiënten
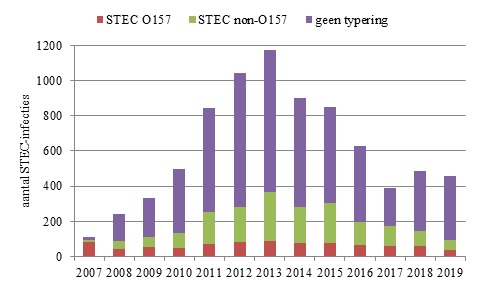
In 2019 werden 459 patiënten met een STEC-infectie gemeld (Figuur 1). Van deze patiënten hadden 35 een bevestigde STEC O157-infectie en 59 een STEC non-O157-infectie. Van de overige patiënten kon in ingestuurde isolaat geen STEC aangetoond worden (n=24) of er was geen isolaat ingestuurd (n=341). Het percentage meldingen van STEC-infecties waarbij geen isolaten werden ingestuurd en dus geen STEC-type kon worden bepaald, is in de afgelopen jaren gestegen van 44% in 2017 naar 64% in 2018 en 74% in 2019.Meer vrouwen (61%) dan mannen hadden een STEC-infectie. HUS werd gemeld bij 22 patiënten, waarvan 12 jonge kinderen (0-6 jaar), 3 oudere kinderen/tieners (9-16 jaar) en 7 volwassenen (21-81 jaar). Bij 7 HUS-patiënten was de O-typering bekend: 2x O157 (2 jonge kinderen) en 4x O26 (3 jonge kinderen en 1 volwassene) en 1x O113 bij 1 volwassene. Bij 2 van de 15 HUS-patiënten zonder bevestigde STEC-infectie werd wel een E. coli zonder stx-genen gevonden: 1x een O26 (jong kind) en 1x een O113 (volwassene). Een volwassen vrouw met HUS (O-typering onbekend) overleed als gevolg van de STEC-infectie.
Figuur 1. Aantal STEC-infecties gemeld over de jaren 2007-2019 onderverdeeld naar STEC O157, non-O157, en O-typering niet bekend. Tot 2007 was alleen STEC O157 meldingsplichtig, in juli 2016 zijn de meldingscriteria aangescherpt
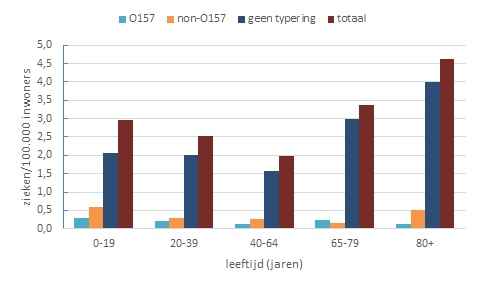
Figuur 2. Incidentie per leeftijdsgroep in 2019 onderverdeeld naar STEC O157, non-O157, O-typering niet bekend en alle STEC-infecties
De meeste STEC O157-infecties (83%) werden in 2019 in de maanden juni tot en met september gezien. In dezelfde periode had 53% van de patiënten een STEC non-O157-infectie of een niet-getypeerde STEC-infectie. De mediane leeftijd van de patiënten varieerde van 27 jaar (O157) en 28 jaar (non-O157) tot 47 jaar (niet getypeerd) (tabel 1). De incidentie van STEC-infecties onder 0-19 jarigen was 3,0 per 100.000 inwoners; dit aantal was lager in de leeftijdsgroepen 20-39 en 40-64 jaar, maar steeg in de hoogste 2 leeftijdsgroepen (figuur 2). De incidentie van STEC O157-infecties leek redelijk stabiel over de leeftijdsgroepen, terwijl STEC non-O157-infecties meer in de laagste en hoogste leeftijdsgroepen leken voor te komen. Meer dan 95% van de patiënten werd gemeld met in ieder geval diarree (tabel 1). Patiënten met een STEC O157-infectie hadden beduidend meer klachten van bloed in de ontlasting. misselijkheid en buikpijn/kramp en werden beduidend vaker in het ziekenhuis opgenomen dan patiënten met andere STEC-infecties. In 12% (4-23%) van de gevallen werd melding gemaakt van andere zieken in de omgeving van de patiënt. Dit waren voornamelijk gezins-/huishoudcontacten. In 27-29% van de gevallen was de infectie in het buitenland opgelopen.
Tabel 1. Kenmerken van STEC-infecties gemeld in 2019
| O157 | STEC non- O157 | Niet getypeerd | ||
| N | 34 | 59 | 365 | |
| mediane leeftijd (range) | 27(1-82) | 28(1-86) | 47 (0-93) | |
| geslacht - vrouw (%) | 17/35(49) | 33/59 (56) | 230/365 (63) | |
| diarree (%) | 34/35(97) | 59/59 (100) | 344/362 (95) | |
| bloed in ontlasting (%) | 21/35(60) | 19/59 (32) | 136/362 (38) | |
| braken (%) | 11/35(31) | 14/59 (24) | 98/362 (27) | |
| misselijk (%) | 18/35(51) | 19/59 (32) | 100/362 (28) | |
| buikpijn (%) | 28/35 (80) | 34/59 (58) | 176/362 (49) | |
| buikkramp (%) | 19/35 (54) | 20/59 (34) | 142/362 (39) | |
| minder urineren (%) | 3/35 (9) | 7/59 (12) | 22/362 (6) | |
| koorts (%) | 7/35/ (20) | 8/59 (14) | 75/362 (21) | |
| ziekenhuisopname (%) | 13/35 (37) | 14/57 (25) | 106/359 (30) | |
| HUS (%) | 2/35 (6) | 5/59 (8) | 15/365 (4) | |
| overleden (%) | 0 | 0 | 1/363 (0,3) | |
| andere zieken in omgeving (%) | 7/35 (23) | 2/53 (4) | 39/309 (13) | |
eigen gezin/ huishouden | 6 | 2 | 30 | |
| overig | 2 | 0 | 9 | |
| buitenland (%) | 10/35 (29) | 15/56 (27) | 96/353 (27) | |
| Afrika | 5 (50) | 6 (32) | 45 (40) | |
| Azië | 1 (10) | 1 (5) | 12 (11) | |
| Europa | 4 (40) | 6 (32) | 24 (21) | |
| Overig | 0 (0) | 2 (11) | 15 (13) |
Bij 92%, 84% en 87% van respectievelijk STEC O157-, STEC non-O157- en niet-getypeerde STEC-infecties die in Nederland waren opgelopen of waarvan onbekend was waar de infectie was opgelopen, werd 1 of meer van vragen over risicofactoren ingevuld (tabel 2). Het aantal ingevulde vragenlijsten over patiënten met STEC O157- en non-O157-infecties was beperkt, gezien het aantal meldingen. Het percentage patiënten dat rauw of niet-gaar vlees had gegeten, was met bijna 50% het hoogst in de groep patiënten met een STEC O157-infectie. Rauwe melk was het meest gedronken door patiënten met een STEC non-O157-infectie (20%). Er was weinig verschil in het contact met dieren tussen de 3 patiëntgroepen (50-63%). De verschillen in medicijngebruik en onderliggend lijden waren ook relatief klein: de grootste verschillen waren te zien in het gebruik van immunosuppressiva (13% in de groep zonder typering versus 5-6%) en in het aantal patiënten met onderliggend lijden (38% in de STEC non-O157-groep versus 48-50%).
Tabel 2. Voedselconsumptie, contact met dieren, medicijngebruik en onderliggend lijden gerapporteerd door STEC-patiënten die niet in buitenland waren geweest, 2019
| O157 | STEC non-O157 | Niet getypeerd | |
| Gegevens beschikbaar | 22/24 (92) | 37/44 (84) | 233/269 (87) |
| Vleeseter | 20/22 (91) | 34/36 (94) | 198/215 (92) |
| Biefstuk | 0/17 (0) | 10/29 (34) | 33/174 (19) |
| Tartaar | 1/17 (6) | 3/29 (10) | 9/174 (5) |
| Hamburger | 6/17 (35) | 8/30 (27) | 50/174 (29) |
| Filet américain | 2/17 (12) | 3/29 (10) | 41/174 (24) |
| Carpaccio | 3/17 (18) | 2/29 (7) | 14/174 (8) |
| Rosbief | 1/17 (6) | 3/29 (10) | 12/174 (7) |
| Ossenworst | 2/17 (12) | 0/29 (0) | 9/174 (5) |
| Rauw of niet-gaar vlees | 8/17 (47) | 9/29 (31) | 70/174 (40) |
| Kiemgroenten | 4/20 (20) | 3/31 (10) | 29/186 (16) |
| Slasoorten | 13/20 (65) | 17/31 (55) | 121/186 (65) |
| Rauwe melk | 1/18 (6) | 6/30 (20) | 17/187 (9) |
| Rauwmelkse kaas | 0/18 (0) | 4/30 (13) | 11/187 (6) |
| Contact met dieren | 10/16 (63) | 17/34 (50) | 117/203 (58) |
| Contact met huisdieren | 8/16 (50) | 16/34 (47) | 105/202 (52) |
| Contact met landbouwhuisdieren | 1/16 (6) | 4/34 (12) | 17/201 (8) |
| Contact met mest | 0/17 (0) | 1/34 (3) | 22/207 (11) |
| Immunosuppresiva | 1/20 (5) | 2/34 (6) | 25/197 (13) |
| Maagzuurremmers | 5/20 (25) | 6/34 (18) | 38/197 (19) |
| Cholesterolverlagers | 2/20 (10) | 3/34 (9) | 26/197 (13) |
Onderliggende ziekten | 10/21 (48) | 13/34 (38)
| 105/210 (50) |
Mogelijke bronnen van infectie
In opdracht van de NVWA onderzocht WFSR in 2019 binnen 20 monitoringsprojecten voedsel op de aanwezigheid van STEC.
Vlees
- Net als in 2017 en 2018, werd veel STEC gevonden in retail vlees van kleine herkauwers: vers vlees, vooral lamsvlees (32 isolaten/214 monsters, 15,0%) en vleesbereidingsproducten (4 isolaten/29 monsters, 13,8%);
- Het percentage STEC-positieve monsters van retail gehakt, - vleesbereidingen (10 isolaten/ 308 monsters, 3,2%) en - kalfsvlees (8 isolaten/211 monsters, 3,8%) was lager dan in 2018 (respectievelijk 6,8 en 6,0%);
- Het percentage STEC-positieve monsters van geïmporteerd exotisch vlees was hetzelfde als in 2018 (4 isolaten/49 monsters, 8,2%);
- Zowel bij rauw vlees voor consumptie als vers rundvlees was het percentage positieve monsters minder dan 3% (respectievelijk 4 isolaten/290 monsters, 1,4% en 7 isolaten/286 monsters, 2,4%).
Groente
In 2019 werd extra aandacht besteed aan het monitoren van groente.• twee monsters waren positief: 1 van een maaltijdsalade (1 isolaat/264 monsters en 1 van rauw te consumeren groente (1 isolaat/ 819 monsters);• Alle monsters van wokgroente (280), superfoods (263), mosselen/oesters (45) en zeewier (51) waren negatief voor STEC;• Een partij geïmporteerde verse kruiden testte positief (46 partijen getest).
Landbouwhuisdieren en mest
- Het monitoren van vleeskuikens is in 2019 afgerond, Hierbij zijn 2 positieve monsters gevonden (bij hetzelfde bedrijf) op een totaal van 326 monsters (5 of 6 monsters per bedrijf).
- Het monitoren van varkens is gestart in 2019. Er zijn 49 positieve monsters gevonden op 85 bedrijven (1 monster en 1 isolaat per bedrijf).
In 2019 is er geen onderzoek uitgevoerd naar STEC naar aanleiding van een klacht en/of een vermoede voedselbron van infectie.
In totaal waren in 2019 74 voedselisolaten positief voor STEC, met 32 verschillende O-groepen. Hierbij werd serotype O146:H21 (n=11) het meest gevonden, gevolgd door O113:H21 (n=6) en O113:H4 (n=4). In slechts 1 isolaat werd ook het eae-gen gevonden, een O5-isolaat uit kalfsgehakt. Het serotype van de 2 isolaten van het vleeskuikenbedrijf waren O24:H18. Bij varkens waren O100:H30 (n=12) en O8:H9 (n=9) de meest gevonden serotypen.
Patiëntisolaten
Het RIVM ontving 865 isolaten van 390 patiënten voor typeringsonderzoek. De isolaten van 141 patiënten konden gekoppeld worden aan gemelde infecties (35 STEC O157, 59 STEC non-O157, 24 niet-bevestigde STEC en 23 waarvan de melding is gewist). De isolaten van de overige 249 patiënten konden niet gekoppeld worden en zijn verder buiten de surveillance gelaten, evenals de gewiste meldingen.Van alle 35 STEC O157-patiënten was een isolaat beschikbaar, waarvan 46% beide stx-genen bevatte en 54% bevatte alleen het stx2-gen. Alle O157-isolaten bevatten het eae- en het EHEC-hly-gen. Van de 59 beschikbare STEC non-O157-isolaten bevatten 16 (27%) isolaten alleen het stx1-gen, 31 (53%) isolaten alleen een stx2-gen (waarvan 8 (14%) het stx2f-gen) en 12 (20%) isolaten bevatten beide genen. In totaal hadden 30 (51%) isolaten het eae- en het EHEC-hly-gen, 10 (17%) isolaten hadden alleen het eae-gen, 8 (14%) isolaten alleen het EHEC-hly-gen en 11 (19%) isolaten geen van beide genen. Binnen STEC non-O157 werd STEC O26 (n=17; 29%) het meest gevonden, gevolgd door STEC O63 (n=5; 8%) en STEC O103 en STEC O111 (beide n=4; 7%). In totaal werden er 25 verschillende O-groepen gevonden, naast 2 niet-typeerbare STEC’s. Over de gehele periode 2007-2019 waren na O157 (n=853), O26 (n=251), O91 (n=142), O103 (n=118), O146 (n=105) en O63 (n=101) de 5 meest gevonden O-groepen.
WGS-clusters
Binnen de STEC O157-isolaten van 2019 werden 4 clusters gezien met 2 tot en met 4 isolaten per cluster.
- Het grootste cluster van 4 isolaten bestond uit 3 mannen en 1 vrouw in de leeftijd 15-25 jaar, verspreid over 2 regio’s, die allemaal in een periode van 12 dagen ziek werden. Hoewel de informatie uit de melding geen aanwijzingen gaf over een gezamenlijke bron, is het wel waarschijnlijk dat deze er was;
- Het tweede cluster, met 2 patiënten uit 2019 met 6 weken tussen de eerste ziektedagen en 1 patiënt uit 2017, lijkt een bron in Turkije te hebben gehad, omdat alle 3 patiënten gedurende (een deel van) de incubatieperiode in Turkije waren geweest;
- Het derde cluster bestond uit 3 patiënten met 3 dagen en 2,5 maand tussen de eerste ziektedagen; 1 patiënt was gedurende een deel van de incubatieperiode in Spanje geweest;
- Het vierde cluster bestond uit 2 patiënten die 2 weken na elkaar ziek waren geworden. In de melding waren geen verdere duidelijke overeenkomsten te zien.
Daarnaast waren er 4 clusters waarbij 1 isolaat uit 2019 met minimaal 6 maanden tussen de eerste ziektedagen clusterde met 1 (3 clusters: 2x 2018 en 1x 2017) of 2 isolaten (1 cluster uit 2018) uit voorgaande jaren.
Er was in 2019 1 STEC O26-cluster van 3 patiënten, van wie de eerste ziektedag 2 dagen tot maximaal 3 weken uit elkaar lagen, maar er was verder geen overeenkomst te zien op basis van de beschikbare informatie. Bij STEC O103 waren er 2 isolaten van patiënten die met 2 patiënten uit 2017 en 1 patiënt uit 2018 clusterden zonder verdere aanwijzingen voor een onderlinge link. Ook een STEC O80-isolaat uit 2019 clusterde met een isolaat uit 2018, zonder verdere aanwijzingen voor een onderlinge link. Vier van de 5 STEC O63-patiënten in 2019 behoorden tot het STEC O63-cluster, waartoe ook 2 van de 3 patiënten uit 2018 en alle 7 patiënten uit 2017 behoorden; het isolaat van de vijfde patiënt lag op 9 allelen afstand en is daarom waarschijnlijk niet verwant.
Discussie
De surveillance van STEC is sinds juli 2016 beperkt tot de acute infecties. Deze infecties lenen zich het beste voor (preventieve) GGD-acties tegen verdere verspreiding in de vorm van advies en voorlichting, en geven een beter beeld van eventuele verheffingen dan chronische en/of vage klachten waarbij een STEC is gevonden.
Het aantal meldingen in 2019 was met 459 patiënten vergelijkbaar met 2018 (487 meldingen). Het aantal STEC O157 (n=35) en STEC non-O157 (n=59) infecties lag wel lager dan in 2018 (respectievelijk 59 en 86 meldingen). Er is echter sprake van een discrepantie tussen de laboratoriumsurveillance en de meldingsplicht. Van de 390 personen van wie 1 of meer isolaten voor typering waren ingestuurd kon slechts 36% (n=141, waarvan 23 een gewiste melding betreft) gekoppeld worden aan de meldingen. Het is niet bekend waarom bij de meeste isolaten de melding ontbrak. Een reden zou kunnen zijn dat de patiënt niet aan de meldingscriteria voldeed, zoals diarree/bloed in ontlasting/braken of acute infectie. Binnen de meldingsplicht kon aan 26% van de meldingen een isolaat gekoppeld worden. Dus van de meeste gemelde STEC-patiënten was geen isolaat ingestuurd door het betreffende (regionale) laboratorium.
WGS is een betrouwbare en relatief snelle methode voor clusterdetectie en -bevestiging. (5) Er is in juli 2019 een verzoek naar de laboratoria verstuurd om in ieder geval van patiënten die opgenomen zijn in het ziekenhuis, bloed in ontlasting hebben en/of HUS, een isolaat in te sturen voor typering. Het effect daarvan is nog minimaal, slechts enkele laboratoria lijken hier gehoor aan te hebben gegeven. De belangrijkste reden hiervoor is het gebruik van PCR voor de diagnose, een verdere kweek is hiervoor niet nodig. Deze trend vormt een bedreiging voor de voor de landelijke surveillance omdat clusterdetectie zeer afhankelijk is van de beschikbaarheid van een isolaat. Zonder (voldoende) isolaten is er geen goed landelijk beeld van circulerende typen (wat zeker belangrijk is bij meer ernstige ziekte) en kan er geen adequaat uitbraakdetectie/-onderzoek worden uitgevoerd. Daarnaast worden de humane en niet-humane isolaten gebruikt in attributiestudies (bijvoorbeeld (2)), waarbij door typeringen te vergelijken, inzicht wordt verkregen in de reservoirs van STEC en eventuele verschuivingen daarin. Deze data geven aanknopingspunten voor preventie en bestrijding. Echter, attributiestudies kunnen alleen uitgevoerd worden als er voldoende isolaten beschikbaar zijn.
Sinds 2013 komt STEC O26 op de tweede plaats, na STEC O157, voor wat betreft het aantal infecties in Nederland. Ook in 2019 was STEC O26 met 29% van de getypeerde STEC non-O157-infecties de meest voorkomende en vormde samen met STEC O157 meer dan de helft van alle getypeerde STEC-infecties. Hoewel STEC O26 iets minder voorkwam dan STEC O157 is de infectie qua ernst zeker belangrijk. In 2019 kregen 4 van 17 patiënten (24%) met een STEC O26-infectie, HUS, ten opzichte van 2 van 35 patiënten (6%) met een STEC O157-infectie. In Europa werden in 2018 ook meer HUS-patiënten gemeld met een STEC O26-infectie dan met een STEC O157-infectie (6).
In voedselmonsters werden voornamelijk O146:H21, O113:H21 en O113:H4 gevonden. Onder de patiënten met een STEC-infectie in 2019 waren 2 besmet met O146:H21, 1 met O146:H28, en 3 met O113:H21 waarvan 1 patiënt met HUS. Er werden in 2019 geen WGS-clusters met zowel humane als voedselisolaten gevonden. Recent is een Nederlandse attributiestudie uitgevoerd met gegevens en isolaten van patiënten en runderen, schapen en geiten, varkens en pluimvee uit de surveillance van 2011 tot en met 2014. (2) Daaruit blijkt dat ongeveer de helft van alle STEC-patiënten kan worden toegeschreven aan runderen als reservoir en aan de O-types O157, O26, O91 en O103 zelfs voor 61-75%. Ongeveer een kwart van de infecties kan worden toegeschreven aan schapen en geiten en daarmee aan 71-77% van de STEC O146-infecties. Varkens vormen slechts een klein reservoir van STEC en pluimvee is als bron verwaarloosbaar.
Sinds 2017 zijn zowel van patiëntisolaten- als van voedselisolaten WGS-data beschikbaar. Dit leverde nog geen link op tussen beide groepen. Een van de redenen kan zijn, dat de NVWA steekproefsgewijs test, terwijl een product vaak maar korte tijd (1 of enkele partijen) besmet zal zijn. Het grootste cluster van STEC O157 bestond uit 4 patiënten die allen in een periode van minder dan 2 weken ziek waren geworden. Waarschijnlijk was hier sprake van een uitbraak met een gezamenlijke bron, maar op basis van de beschikbare gegevens werd deze niet gevonden.
In 2017, het eerste jaar met WGS-data, viel op dat STEC O63-isolaten sterk clusterden. Twee van de 3 patiënten uit 2018 en 4 van de 5 uit 2019 vielen ook in dit cluster, de 2 overige patiënten hadden een iets afwijkende stam. Het lijkt erop dat STEC O63-infecties die in Nederland tot nu toe gezien zijn, weinig diversiteit laten zien. STEC O63-isolaten bevatten altijd het stx2f-gen, wat in de meeste gevallen een milde infectie veroorzaakt. (7) Duiven werden als mogelijk reservoir van stx2f-bevattende E.coli’s gezien, maar een vergelijking op basis van WGS-data van isolaten uit duiven en uit patiënten liet zien dat het vaak om verschillende serotypes en aparte subclusters gaat. (8) Het reservoir van humane stx2f-STEC blijft daarmee grotendeels onopgehelderd. Mens-op-menstransmissie zou een belangrijke rol kunnen spelen bij STEC O63-infecties.
Conclusie
STEC O157 en O26 zijn de belangrijkste verwekkers van STEC-infecties in Nederland, zowel in voorkomen als in ernst. In voedsel- en dierisolaten werden voornamelijk STEC O146, O113, O100 en O8 aangetroffen. De focus van de STEC-surveillance ligt sinds juli 2016 op acute infecties. Met de invoering van de nieuwe meldingscriteria is het aantal meldingen gedaald, maar het aandeel meldingen zonder isolaat (of WGS-data) is gestegen naar 74%. Dit vormt een bedreiging voor de surveillance, omdat clusterdetectie zeer afhankelijk is van de beschikbaarheid van isolaten voor STEC-typering en clusteranalyse met behulp van WGS-data. Een groter aantal patiënt- en voedselisolaten zou meer inzicht kunnen geven in bronnen van STEC en mogelijk ook tot meer matches kunnen leiden.
Wij bedanken alle GGD'en en medisch microbiologische laboratoria hartelijk voor hun medewerking bij het verzamelen van de patiëntgegevens en het insturen van isolaten. Ook bedanken we de medewerkers van de NVWA en WFSR voor het bemonsteren van en onderzoek naar landbouwhuisdieren en (verdacht) voedsel.
Auteurs
I.H.M. Friesema (1), S. Kuiling (1), M. van der Voort (2), K. van der Zwaluw (1), M. van den Beld (1), P.H. In ‘t Veld (3), E. Franz (1)
- RIVM, Centrum Infectieziektebestrijding
- Wageningen Food Safety Research (WFSR), Microbiological and chemical food analysis
- Nederlandse Voedsel- en Warenautoriteit (NVWA), Divisie Handhaving
Correspondentie
Literatuur
- Smith J.L., Fratamico P.M., Gunther N.W. Shiga Toxin-Producing Escherichia coli. Advances in applied microbiology 2014; 86: 145-97.
- Mughini-Gras L., van Pelt W., van der Voort M., Heck M., Friesema I., Franz E. Attribution of human infections with Shiga toxin-producing Escherichia coli (STEC) to livestock sources and identification of source-specific risk factors, The Netherlands (2010-2014). Zoonoses Public Health 2018; 65: e8-e22.
- Friesema I.H., Van De Kassteele J., De Jager C.M., Heuvelink A.E., Van Pelt W. Geographical association between livestock density and human Shiga toxin-producing Escherichia coli O157 infections. Epidemiol Infect 2011; 139: 1081-7.
- Derzelle S., Grine A., Madic J., et al. A quantitative PCR assay for the detection and quantification of Shiga toxin-producing Escherichia coli (STEC) in minced beef and dairy products. International Journal of Food Microbiology 2011; 151: 44-51.
- Jenkins C., Dallman T.J., Grant K.A. Impact of whole genome sequencing on the investigation of food-borne outbreaks of Shiga toxin-producing Escherichia coli serogroup O157:H7, England, 2013 to 2017. Euro Surveill 2019; 24.
- EFSA and ECDC. The European Union One Health 2018 Zoonoses Report. EFSA Journal 2019; 17: 5926.
- Friesema I., van der Zwaluw K., Schuurman T., et al. Emergence of Escherichia coli encoding Shiga toxin 2f in human Shiga toxin-producing E. coli (STEC) infections in the Netherlands, January 2008 to December 2011. Euro Surveill 2014; 19.
- van Hoek A., van Veldhuizen J.N.J., Friesema I., et al. Comparative genomics reveals a lack of evidence for pigeons as a main source of stx2f-carrying Escherichia coli causing disease in humans and the common existence of hybrid Shiga toxin-producing and enteropathogenic E. coli pathotypes. BMC Genomics 2019; 20: 271.
Infectieziekten Bulletin, jaargang 31, nummer 3, december 2020