Whole genome sequencing van Mycobacterium tuberculosis
De ultieme tool voor identificatie, gevoeligheidsbepaling en epidemiologische typering?
Tuberculose, bijna altijd veroorzaakt door Mycobacterium tuberculosis, is wereldwijd de meest dodelijke infectieziekte. In Nederland werden in 2018 in totaal 806 tuberculosepatiënten gemeld in het nationaal tuberculoseregister. Hiervan was 77% afkomstig was uit het buitenland, voornamelijk Eritrea, Marokko, India en Somalië.(1) In 2016 is bij het RIVM tuberculosereferentielaboratorium een vierjarig traject gestart voor het invoeren van whole genome sequencing (WGS) van M. tuberculosis complex (MTBC) in de diagnostiek. WGS is een laboratoriumtechniek waarbij het complete genoom (≈ 4.4 miljoen baseparen) van M. tuberculosis in kaart wordt gebracht. Deze techniek wordt bij het RIVM gebruikt naast de routinediagnostiek om de bruikbaarheid van WGS te kunnen vergelijken met de standaardtechnieken.
Het MTBC bestaat uit de (sub)species M. tuberculosis, M. canettii, M. orygis, M. pinnipedii, M. caprae, M. bovis, M. bovis BCG, M. microti, M. mungi, M. suricattae, en de zogenoemde dassie bacillus. M. tuberculosis kan worden onderverdeeld in 7 genotypefamilies. Het herkennen van isolaten is van belang voor epidemiologisch onderzoek en de behandeling. M. bovis bijvoorbeeld is niet gevoelig voor pyrazinamide, een antibioticum dat in de eerstelijn wordt gebruikt om tuberculose te behandelen. M. bovis en M. pinnipedii veroorzaken meestal extra-pulmonale tuberculose en hebben bijna altijd een dierlijke bron. M. pinnipedii wordt vrijwel uitsluitend gevonden bij zeehonden, zeeleeuwen en aanverwante soorten. M. bovis BCG wordt gevonden als complicatie na BCG-vaccinatie en na behandeling van blaascarcinomen met een BCG-preparaat. (2-4) Momenteel wordt voor het herkennen van (sub)species gebruik gemaakt van de reverse-line-blot-methode die gebaseerd is op variaties in het gyrase B-gen en een bepaalde grote genomische deletie. Echter, de reverse-line-blotmethode kan geen onderscheid maken tussen M. tuberculosis en M. canettii en ook niet tussen M. africanum, M. pinnipedii en M.orygis. Daarnaast kan de reverse-lineblotmethode géén van de genotypen van M. tuberculosis herkennen.
De klassieke methode om resistentie tegen antibiotica te vinden is het gebruik van de fenotypische Mycobacterium Growth Indicator Tube (MGIT); dit is tevens de internationale referentiestandaard van de World Health Organization (WHO). MGIT is gebaseerd op het testen van de gevoeligheid van M. tuberculosis bij 1 kritische concentratie van een antibioticum. Naast het feit dat de uitslagen van de MGIT soms weken of maanden op zich laten wachten vanwege de langzame groei van M. tuberculosis, is deze methode niet altijd accuraat en reproduceerbaar:
- Het testen van de gevoeligheid tegen pyrazinamide bijvoorbeeld vergt speciale groeicondities: er wordt een relatief groot inoculum van de bacteriën getest vergeleken met de testen voor andere eerstelijnsmiddelen, wat kan leiden tot fout-positieve uitslagen. Daarnaast kan de remming van de bacteriële groei het gevolg zijn van de zure omgeving waarin pyrazinamide uitsluitend actief is in plaats van daadwerkelijke gevoeligheid van M. tuberculosis.
- Een andere, zeer relevante tekortkoming van MGIT is het feit dat in principe uitsluitend 1 kritische concentratie van een middel wordt getest. Hierdoor is het niet mogelijk om een laag niveau van resistentie te vinden, waardoor de desbetreffende isolaten in de MGIT soms onterecht als gevoelig worden beschouwd. Fout-negatieve resistentiebepalingen kunnen resulteren in een slechte behandeluitkomst en uiteindelijk bij de patiënt zelfs tot een terugval leiden als hij/zij ten gevolge van de negatieve uitslag een te lichte behandeling krijgt. Dit is ongewenst, vooral omdat er aanpassingen in de doseringen van isoniazide en rifampicine mogelijk zijn waarbij de patiënt, in geval van een laag niveau van resistentie, met een hogere dosis behandeld kan worden.
Naast de fenotypische testen werden in de afgelopen decennia ook diverse snelle DNA-methoden ontwikkeld om specifieke resistentiemutaties te vinden. (5,6) Echter, deze sneltesten beperken zich tot een aantal bekende resistentiemutaties terwijl de praktijk weerbarstiger is: er komen veel meer variaties voor van resistenties dan je met dit soort testen kunt vinden.
Sinds 1993 worden alle isolaten van M. tuberculosis naar het tuberculosereferentielaboratorium van het RIVM gestuurd voor epidemiologische typering en, op aanvraag, voor resistentiebepalingen en het herkennen van (sub)species. Voor alle isolaten in de periode 2009-2018 werd gebruik gemaakt van de typeringsmethode Variable Number Tandem Repeat (VNTR). Deze methode is gebaseerd op amplificatie van 24 repeterende loci/regio’s in het genoom, wat resulteert in een numerieke code van 24 getallen die het aantal repeats in elk van de 24 geteste genomische regio’s representeert. Isolaten met een identiek 24-loci VNTR-patroon worden aan een VNTR-cluster toegekend, wat een epidemiologisch verband suggereert tussen de betreffende patiënten. De informatie over de clustering van patiënten op basis van VNTR werd tot en met 2018 wekelijks met GGD’en gedeeld ter ondersteuning van het bron-en contactonderzoek. Het onderscheidende vermogen van VNTR was echter niet optimaal. Ondanks actieve bronopsporing van patiënten, vonden GGD’en in de praktijk bij slechts ≈ 26% van de op basis van VNTR geclusterde patiënten een epidemiologisch verband. (7) Op basis van VNTR was er bijvoorbeeld vaak sprake van clustering tussen asielzoekers en immigranten met verschillende achtergronden die GGD’en niet aan elkaar konden linken.
In dit artikel zullen de gepubliceerde resultaten worden samengevat over het gebruik van WGS voor:
- herkennen van (sub)species en genotypen binnen het MTBC
- de bepaling van de gevoeligheid van MTBC-isolaten voor eerstelijnsmiddelen
- het in kaart brengen van de genetische afstand tussen isolaten om transmissie tussen tuberculose- patiënten te volgen
Identificatie van (sub)species en genotypen binnen het MTBC
In samenwerking met de Oxford Universiteit in Engeland is de op WGS gebaseerde SNP-IT(SNPs to Identify TB)-methode ontwikkeld voor het herkennen van (sub)species en genotypen binnen het MTBC. (8) De nieuwe methode is ontwikkeld op basis van een maximaal gevarieerde set - wat betreft (sub)species en genotype families - van 323 isolaten uit collecties van het RIVM en de Oxford Universiteit. SNP-IT is gebaseerd op meervoudige SNPs (fylogenetische SNPs) die uniek zijn voor de desbetreffende (sub)species of genotypen en die dus afwezig zijn in de overige (sub)species en genotypen. Met SNP-IT werden in totaal 13.893 fylogenetische SNPs geïdentificeerd, waarbij M. canettii het grootste aantal karakteristieke fylogenetische SNPs bevat (n = 6.837) en M. bovis het kleinste aantal (n = 23). SNP-IT werd gevalideerd op 614 sequenties van MTBC-isolaten uit de database van Public Health England en een internationale database. Alle 614 sequenties uit de validatieset werden correct geïdentificeerd met SNP-IT.
De interne validatie bij het RIVM van in totaal 742 sequenties liet 2 afwijkingen zien waarbij de isolaten werden aangeduid als M. orygis met de SNP-IT, maar als M. africanum met de reverse-line-blotmethode. Op basis van pncA-mutaties en de VNTR bleken deze 2 isolaten inderdaad tot M. orygis te behoren en dus correct geïdentificeerd met SNP-IT. Verder werden met de reverse-line-blot-methode 699 isolaten als M. tuberculosis geïdentificeerd, terwijl deze met SNP-IT verder onderverdeeld werden in verschillende genotypen zoals Beijing, Delhi/Central-Asian en Euro-American.
Gevoeligheidsbepaling voor eerstelijnsmiddelen
De betrouwbaarheid van WGS ten opzichte van MGIT om de gevoeligheid van M. tuberculosisisolaten te testen tegen de eerstelijnsmiddelen rifampicine, isoniazide, ethambutol en pyrazinamide werd onderzocht in een populatiestudie. (9) Met WGS werden 9 genen en/of promotorregio’s onderzocht op mutaties die geassocieerd zijn met resistentie. De isolaten die niet overeenkomstige resultaten gaven in de MGIT/WGS werden opnieuw getest in de MGIT door de kritische concentratie en een concentratie net onder het breekpunt te testen. Verder werd de incubatietijd in de MGIT verlengd tot 45 dagen. De laatste 2 aanpassingen werden toegepast om ook laagresistente isolaten te vinden. De voorspellende waarden van WGS werden vergeleken met de MGIT-referentiestandaard én met een zogenoemde samengestelde referentiestandaard. Isolaten met (laag) resistentiegeassocieerde mutaties voor rifampicine, isoniazide en ethambutol die vaak als gevoelig worden gerapporteerd met MGIT, maar die een verhoogde minimaal remmende concentratie lieten zien bij het hertesten met MGIT, werden als resistent ingevoerd in de samengestelde referentiestandaard.
Deze studie omvatte 1.121 MTBC-isolaten (met uitzondering van M. bovis BCG) uit 2016 en 2017, waarvan ≈ 11% resistent was tegen 1 of meerdere antibiotica. De vergelijking van WGS- met de initiële MGIT-resultaten toonde 57/1.121 (5.1%) afwijkende isolaten. De meeste afwijkingen voor isoniazide en ethambutol konden verklaard worden doordat deze bacteriële groei lieten zien bij de concentraties net onder het breekpunt wanneer ze opnieuw getest werden met MGIT. De meeste afwijkingen voor rifampicine waren te verklaren doordat deze bacteriële groei vertoonden bij langere incubatie in de MGIT bij het hertesten. Beiden zijn aanduidingen voor een laag niveau van resistentie die normaal bij het testen van 1 breekpunt als ‘gevoelig’ werden gerapporteerd met MGIT. De meeste afwijkingen voor pyrazinamide waren te wijten aan het algemeen bekende probleem van de lage reproduceerbaarheid van de MGIT-gevoeligheidsbepaling voor dit antibioticum. De negatief voorspellende waarde van WGS ten opzichte van de MGIT-referentiestandaard was 100%, 99.8%, 99.9% en 99.3% voor respectievelijk rifampicine, isoniazide, ethambutol en pyrazinamide (tabel 1). Dit houdt in dat wanneer met WGS geen mutaties in de resistentiegenen werden gevonden, dit in ≥ 99.3% van de gevallen ook MGIT-gevoelige isolaten waren. De positief voorspellende waarde van WGS ten opzichte van de samengestelde referentiestandaard steeg aanzienlijk naar 91.2%, 99% en 80% voor respectievelijk rifampicine, isoniazide en ethambutol, wat de tekortkomingen van de MGIT als referentiestandaard weergeeft voor het aantonen van laagresistente isolaten (tabel 1).
Tabel 1. Voorspellende waarden van WGS ten opzichte van de MGIT-referentiestandaard en de samengestelde referentiestandaard voor de 1.121 MTBC-isolaten
| MGIT-referentiestandaard | Samengestelde referentiestandaard | |||||||
sensiti- viteit | specifi- citeit | PVW | NVW | sensiti- viteit | specifi- citeit | PVW | NVW | |
| Rifampicine | 100% | 99.2% | 73.5% | 100% | 96.6% | 99.7% | 91.2% | 99.9% |
| Isoniazide | 97.8% | 99.2% | 91.9% | 99.8% | 98% | 99.9% | 99% | 99.8% |
| Ethambutol | 80% | 98.6% | 20% | 99.9% | 94.1% | 99.6% | 80% | 99.9% |
| Pyrazinamide | 85.1% | 98.8% | 75.5% | 99.3% | - | - | - | - |
| PVW=positief voorspellende waarde; NVW=negatief voorspellende waarde | ||||||||
Epidemiologische typering
Op alle MTBC-isolaten uit 2016 (10) en 2017 werd zowel de VNTR-typering als WGS toegepast. Isolaten met een identiek 24-loci VNTR-patroon werden als ‘geclusterd’ beschouwd en de patiënten in deze clusters werden doorgegeven aan GGD’en als ‘mogelijk epidemiologisch met elkaar verbonden’. De definitie van clustering van isolaten op grond van WGS was een maximale genetische afstand van 12 SNPs tussen isolaten.
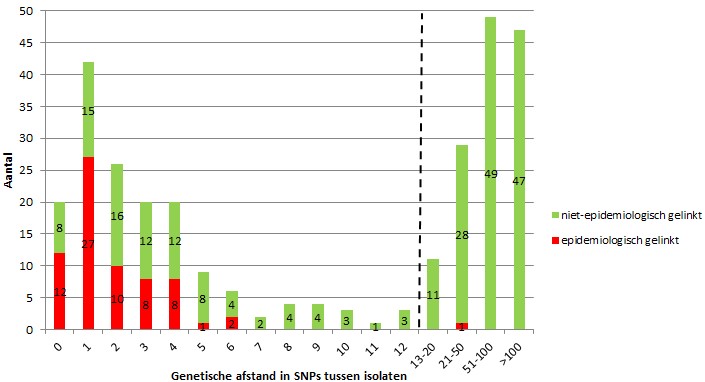
In totaal werden 1.108 isolaten getypeerd met zowel WGS als VNTR, waarvan 427 (39%) clusterden op basis van VNTR en 277 (25%) op basis van WGS. Brononderzoek door GGD’en naar de 427 VNTR- geclusterde patiënten resulteerde in 110 bevestigde epidemiologische verbanden. De genetische afstanden tussen de 427 VNTR-geclusterde isolaten staan in figuur 1. Onder de WGS-geclusterde patiënten werden 108/110 epidemiologische verbanden gevonden. Eén epidemiologisch verband tussen 2 patiënten werd niet gevonden met WGS; volgens de GGD was er voor de vervolgpatiënt risico geweest op blootstelling tijdens de besmettelijke periode van de bronpatiënt, maar beide patiënten waren < 2.5 jaar in Nederland en geboren in landen die hoog endemisch zijn voor tuberculose. Het is mogelijk dat deze patiënten door een andere bron waren geïnfecteerd tijdens de vluchtroute naar Nederland of dat zij al waren geïnfecteerd in het land van herkomst.
Figuur 1. Genetische afstand in SNPs tussen de 427 VNTR-geclusterde isolaten uit 2016 en 2017
Discussie/conclusie
De betrouwbaarheid van WGS voor het herkennen van (sub)species en genotypen, het aantonen van de gevoeligheid tegen eerstelijnsmiddelen en de epidemiologische typering van MTBC-isolaten is groter dan bij de conventionele laboratoriumtechnieken. WGS heeft in de typering een veel hoger onderscheidend vermogen dan VNTR om epidemiologische verbanden op te sporen. Met WGS daalt het aantal geclusterde patiënten aanzienlijk, terwijl alle epidemiologische verbanden wel degelijk worden gevonden. Dit betekent dat GGD’en op een nog efficiëntere wijze bron-en contactonderzoek kunnen uitvoeren. De validatie bij het RIVM en in Engeland geven aan dat de nieuwe op WGS gebaseerde SNP-IT-methode een uiterst betrouwbare benadering is en specifieker is dan de huidige methode, omdat met SNP-IT, naast alle (sub)species, ook de genotypen herkend worden. Daarnaast heeft WGS een zeer hoge negatief voorspellende waarde (≥ 99.3%) voor het voorspellen van gevoeligheid tegen de eerstelijnsmiddelen rifampicine, isoniazide, ethambutol en pyrazinamide, door het uitsluiten van aanwezigheid van resistentiemutaties. De positief voorspellende waarde verschilt echter sterk per mutatie en zal ook als dusdanig worden gerapporteerd. Verder kan met WGS op accurate wijze een laag niveau van resistentie tegen antibiotica gevonden worden. Dit wordt soms met MGIT gemist omdat isolaten onterecht als ‘gevoelig’ worden getest omdat dat slechts 1 kritische concentratie van het antibioticum in de MGIT-bepaling getest wordt.
In Engeland gebruikt men WGS sinds kort voor het herkennen van (sub)species/genotypen, de screening op gevoeligheid en de epidemiologische typering van M. tuberculosis. In Nederland gebruikt men vanaf januari 2019 WGS voor de typering en zal de overstap voor het herkennen van (sub)species/genotypen en het screenen op de gevoeligheid voor antibiotica, in 2020 worden gemaakt. Vanzelfsprekend blijven de fenotypische testen uitgevoerd worden met uitgebreidere concentratiereeksen als er resistentiemutaties worden aangetoond, of op speciaal verzoek.
Al met al is de invoering van WGS in de laboratoriumdiagnostiek van tuberculose een aanzienlijke stap voorwaarts. Op dit moment is de beschreven techniek toepasbaar op positieve MGIT-kweken. Er wordt echter al gewerkt aan het direct toepassen van WGS op klinisch materiaal, waarbij momenteel helaas de onnauwkeurigheid nog een groot probleem vormt vanwege de aanwezigheid van humaan DNA en overig niet-mycobacterieel DNA in de sequenties. Het is wel mogelijk om targeted sequencing toe te passen op klinisch materiaal. Hierbij worden genomische regio’s van interesse, zoals de resistentiegenen, duizenden malen vermenigvuldigd, waardoor de gevoeligheid van de test sterk toeneemt. Hoewel uit deze targeted sequencing minder informatie verkregen kan worden dan uit WGS, wordt dit in de komende periode een belangrijke toepassing in regionale en perifere laboratoria.
Auteurs
R. Jajou, D. van Soolingen, Centrum Infectieziektebestrijding, Rijksinstituut voor Volksgezondheid en Milieu, Bilthoven
Correspondentie
rana.jajou@rivm.nl of dick.van.soolingen@rivm.nl
Literatuur
- RIVM. Tuberculosis keypoints the Netherlands 2018. Beschikbaar via: https://www.rivm.nl/sites/default/files/2019-03/011226_120101_Kerncijfer....
- Lamm DL, van der Meijden PM, Morales A, et al. Incidence and treatment of complications of bacillus Calmette-Guerin intravesical therapy in superficial bladder cancer. J Urol. 1992;147(3):596-600.
- Elkabani M, Greene JN, Vincent AL, et al. Disseminated Mycobacterium bovis after intravesicular bacillus calmette-Gu rin treatments for bladder cancer. Cancer Control. 2000;7(5):476-81.
- Bakker M, Stadhouders NAM, Van Hest NAH, et al. Tuberculose door het BCG-vaccin. Tegen de Tuberculose. 2013;109(3):8-9.
- Boehme CC, Nabeta P, Hillemann D, et al. Rapid molecular detection of tuberculosis and rifampicin resistance. N Engl J Med. 2010;363:1005-15
- Schön T, Miotto P, Koser CU, et al. Mycobacterium tuberculosis drug-resistance testing: challenges, recent developments and perspectives. Clin Microbiol Infect. 2017;23:154-60.
- Slump E, Blijboom L, Bregman IM, et al. Tuberculose in Nederland 2017: Surveillancerapport inclusief rapportage monitoring van interventies. 2018. Beschikbaar via: https://www.rivm.nl/bibliotheek/rapporten/2018-0143.pdf.
- Lipworth S, Jajou R, de Neeling A, et al. SNP-IT Tool for Identifying Subspecies and Associated Lineages of Mycobacterium tuberculosis Complex. Emerg Infect Dis. 2019;25(3):482-8.
- Jajou R, van der Laan T, de Zwaan R, et al. WGS more accurately predicts susceptibility of Mycobacterium tuberculosis to first-line drugs than phenotypic testing. J Antimicrob Chemother. 2019: doi:10.1093/jac/dkz215.
- Jajou R, de Neeling A, van Hunen R, et al. Epidemiological links between tuberculosis cases identified twice as efficiently by whole genome sequencing than conventional molecular typing: A population-based study. PLoS One. 2018;13(4):e0195413.
Infectieziekten Bulletin, jaargang 30, nummer 6, november 2019